Considérant que les maladies infectieuses, en particulier celles provenant de sources zoonotiques, contribuent actuellement à environ 20% des causes annuelles mondiales de décès et à 10% de la charge totale de morbidité sur le continent européen [1]; et que les pertes probables d’une épidémie de grippe pandémique pourraient atteindre 3 billions de dollars américains (environ 5% du produit intérieur brut mondial) [2], les différents États de l’Union européenne ont décidé de donner la priorité à des domaines de santé comprenant une surveillance proactive et une réponse réactive, en mettant l’accent sur la résistance aux antimicrobiens (RAM), les maladies évitables par la vaccination, la tuberculose, la grippe et les infections sexuellement transmissibles.
À cet égard, il est crucial de comprendre la voie qui conduit les données à passer de manière réaliste des laboratoires cliniques à des logiciels de surveillance intégrée très avancés, comme c’est le cas pour les agences gouvernementales nationales (par exemple, Public Health England). Le programme de surveillance des maladies commandé par Public Health England (PHE) couvre 37 maladies infectieuses, dont la grippe.
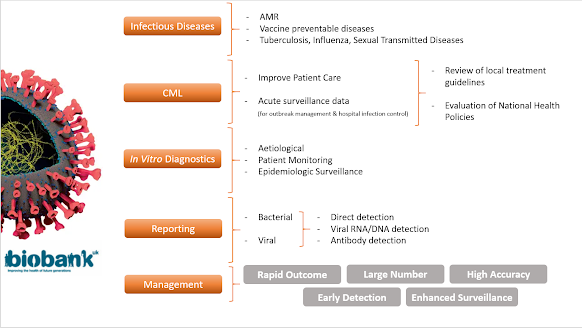
Les laboratoires de microbiologie clinique (LMC) démontrent leur capacité à ::
o informer et améliorer les soins individuels aux patients,
o contribuer à la gestion des éclosions et au contrôle des infections à l’hôpital;
o fournir des données de surveillance précises sur les maladies infectieuses et la RAM. Ces informations peuvent ensuite être utilisées pour l’examen des directives de traitement locales et la conception et l’évaluation des politiques nationales de santé. [1]
En ce sens, In vitro diagnostic jouer un rôle important dans le processus de contrôle – Il y a au moins trois grands domaines où in vitro les diagnostics peuvent apporter des contributions essentielles au raisonnement diagnostique et à la prise en charge des patients atteints d’une infection suspectée ou confirmée:
o diagnostic étiologique,
o surveillance des patients,
o et la surveillance épidémiologique. [3]
Avec un intérêt particulier pour le Rdéport d’infections bactériennes et virales – En général, nous pouvons séparer la détection des virus en trois catégories principales:
o détection directe du virus,
o détection d’ARN/ADN viral, et
o détection d’anticorps. [4]
Les rapports de données alimentent deux systèmes de surveillance très importants, la Biobanque britannique (UKB) (une ressource de santé internationale avec 500K sujets permettant la recherche sur les déterminants génétiques et du mode de vie des maladies courantes), ainsi que le Système de Surveillance de Deuxième Génération (SGSS) de Santé Publique Angleterre (une base de données centralisée de microbiologie couvrant les laboratoires de diagnostic clinique anglais) participent à la surveillance nationale des infections à déclaration obligatoire hautement pertinentes, des isolations bactériennes et de la résistance aux antimicrobiens. [5]
L’analyse des données partagées introduites dans ces systèmes permet une meilleure gestion des scénarios épidémiologiques pertinents basés sur:
o Détection précoce rapide validée par un grand nombre d’échantillons avec un diagnostic de grande précision soutenant une surveillance renforcée.
Et ce processus aidera en synergie le:
o Examen des directives de traitement locales, et
o Évaluation des Politiques Nationales de Santé
Il existe de nombreux défis associés aux rapports de laboratoire au sein du système de surveillance de la santé publique:
o Artefacts de données – données manquantes, incohérentes et invraisemblables; lacunes dans la transmission des données;
o Changement constant des directives et des instructions – indicateurs en évolution rapide;
o Procédures hétérogènes – différences systématiques entre les laboratoires et dans l’utilisation des services; liens avec les patients dans le temps, sources de données différentes, nombreux registres et indicateurs,
o Charge de temps pour les fournisseurs occupés
o Rapports idiosyncratiques +/- – même si la déclaration de faux positifs/négatifs est associée à (A) une faible concentration d’anticorps habituellement présents dans les échantillons fluidiques; (B) la présence de protéines homologues; et (C) le manque de sensibilité de l’instrument de détection, le fait que tous les négatifs ne sont pas signalés et que tous ne le sont pas via les mêmes systèmes de déclaration et aux mêmes agences de surveillance augmente les difficultés de surveillance associées au processus. [3] [4]
o Manque de ressources pour les tests à haut volume / fréquence
o Expédition incongrue vers des laboratoires de référence
o Incompatibilité Numérateur-Dénominateur – une distorsion systématique due à un dénominateur qui ne correspond pas au numérateur, ou vice versa.
o Difficultés d’évaluation de l’exposition
o Environnement/ Équipement Inadéquat/ Insuffisant
o Culture de la cartographie laxiste (par exemple, ISO15189, ISO17025) – Les normes réglementaires exigent que les laboratoires cliniques établissent et documentent leurs propres directives de performance pour les tests développés en laboratoire afin de s’assurer que les résultats obtenus sont effectués avec précision et précision, même avant la mise en œuvre du test. Les aspects pertinents à prendre en compte sont: l’exactitude, la précision, la plage à déclarer, l’intervalle de référence, la sensibilité analytique et la spécificité analytique.
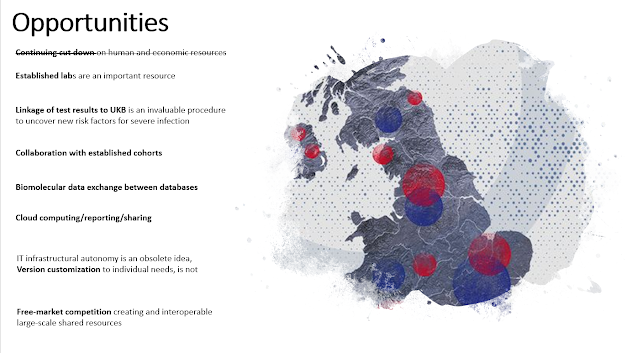
• La première et peut-être la plus importante leçon que les décideurs politiques et les administrateurs d’hôpitaux DOIVENT tirer de la COVID-19 est la suivante: la réduction continue des ressources humaines et économiques a un impact énorme sur le bon fonctionnement des structures cela peut facilement conduire à l’effondrement du système de santé public / privé, y compris des laboratoires cliniques avec leur demande accrue de tests. [3]
• Les laboratoires établis sont une ressource importante
• Le lien entre les résultats des tests COVID-19 et l’UKB fournit une ressource inestimable à la communauté de recherche internationale cela a le potentiel de découvrir de nouveaux facteurs de risque d’infection grave. UKB est l’une des cohortes les plus importantes et les plus étudiées au monde. [5]
• Échange de données biomoléculaires entre bases de données – Comme il est si bien indiqué dans Lenert et Sundwall (2012) « Les fournisseurs cliniques doivent échanger des types précis de données avec le système de santé publique, telles que les données sur la vaccination et la surveillance syndromique et la déclaration des maladies à déclaration obligatoire. Cependant, une crise se profile parce que les systèmes de technologie de l’information de la santé publique n’ont en grande partie pas les capacités nécessaires pour accepter les types de données proposées pour l’échange. Le cloud computing peut être une solution pour les systèmes d’information de santé publique. Grâce à des ressources informatiques partagées, les services de santé publique pourraient profiter des avantages de la déclaration électronique« . [6]
• Autonomie des infrastructures informatiques – Il est compréhensible que chaque organisation souhaite conserver son autonomie, cependant et pour une intervention positive rapide, cela est totalement irréalisable dans un système diasporique aux multiples facettes. L’idée est même considérée comme OBSOLÈTE[6] et va à l’encontre de l’inflexion technologique majeure dictée par des services cloud » démocratiques » et très fonctionnels.
• « Mise en place d’un réseau efficace de laboratoires cliniques régionaux Une solution réalisable, à condition qu’une réglementation simple pour le transport des échantillons et la biosécurité soit établie et surveillée, concerne celles qui ne sont pas directement mises en cause par l’épidémie et où les échantillons peuvent être transportés. Ceci, à son tour, met en évidence la nécessité inévitable de déployer des efforts importants pour permettre une meilleure et plus large harmonisation des résultats et des informations de laboratoire, englobant à la fois les questions analytiques et extra-analytiques. » [3]
• Communication efficace avec les parties prenantes appropriées – « Il est essentiel que le personnel du laboratoire soit chargé de communiquer les résultats des tests aux parties prenantes appropriées (c’est-à-dire aux personnes officiellement chargées de faire face à l’épidémie), évitant ainsi de diffuser des informations susceptibles de générer une panique injustifiée, ou une assurance inappropriée, parmi la population générale. » [3]
• Diagnostic précis VS Stigmatisation du patient – « La réalisation, le maintien et l’amélioration de la précision, de la rapidité et de la fiabilité des résultats des tests sont des résultats clés des laboratoires de diagnostic. Les résultats tardifs ou faussement négatifs du test SARS-CoV-2 entraîneront des retards ou même empêcheront un diagnostic correct, mettant en péril l’isolement en temps opportun et la prévention de la transmission. À leur tour, les tests faussement positifs gaspilleront les ressources de santé publique, conduiront à des données épidémiologiques incorrectes et pourraient même conduire à la stigmatisation des patients. Le contrôle de la qualité est la pierre angulaire d’un diagnostic sûr, cohérent et fiable, et de nombreuses études et cadres décrivent la structure des systèmes de gestion de la qualité adaptés aux laboratoires de diagnostic. »[7]
• Intégration dans les bio-dépôts – « Le rôle important des réseaux de LMC ne doit pas être sous-estimé dans le partage des métadonnées cliniques de routine ou des données collectées. Leur intégration potentielle dans un ensemble de données commun (biorépositoires — tel que proposé par le Consortium des normes d’échange de données cliniques) maximiserait les possibilités de traduire les contributions des patients en solutions thérapeutiques et diagnostiques. Le processus de consolidation, par exemple, offre une opportunité tangible d’étendre la portée des analyses groupées de données de biomarqueurs de patients individuels provenant de plateformes et de cohortes de laboratoires hétérogènes à des études au niveau de la population utilisant des algorithmes de fusion. » [1]
• « La disponibilité de kits de diagnostic commerciaux dans les centres périphériques doit faire partie de la stratégie d’identification précoce et précise du plus grand nombre possible de patients infectés. » [3]
• Localement – « Les tests à proximité du patient comprendraient des tests d’amplification d’acides nucléiques dits“plug-and-play” de 1 à 2 h pour lesquels un résultat rapide peut avoir un impact direct sur les soins aux patients. Des tests centralement plus complexes / à volume élevé seraient expédiés dans une installation centrale. En outre, la capacité des CMLS en réseau à accéder à plusieurs partenaires, régions géographiques et spécialités cliniques différentes peut améliorer leurs capacités à fournir une surveillance avancée des maladies et une reconnaissance précoce des épidémies. » [1]
• SOP harmonisées – « Des professionnels de laboratoire peuvent également être mis à disposition sur place, où ils pourraient aider à définir des procédures opérationnelles normalisées (SOP) pour la collecte et le transport des échantillons. Le choix entre ces solutions possibles dépendra évidemment de nombreuses questions économiques, législatives, juridiques, logistiques, environnementales et techniques. » [3]
• Analyses directes le jour même – « Un avantage majeur du CMLs consolidé est l’élargissement de la gamme d’activités, capable de s’adapter à des tests de haute technologie et sophistiqués avec une sensibilité et une spécificité accrues (30), tandis que la couverture quotidienne habituelle est étendue par un deuxième (et un troisième) quart de travail. Le jour même, des essais directs, y compris des essais moléculaires pour des organismes sélectionnés, sont effectués de manière routinière, réduisant ainsi le temps nécessaire pour obtenir des résultats. » [1]
• Flexibilité inhérente du système – « La disponibilité de quantités accrues de données haute résolution à moindre coût crée une anticipation, une exigence et un coût en aval pour l’hébergement, les analyses et l’interprétation de ces données. La flexibilité systémique inhérente qui est nécessaire pour recevoir différents types de données à différentes vitesses et à partir de différents endroits — et relier tout cela aux données cliniques collectées de manière routinière et faire rapport — n’est pas une tâche insignifiante en soi. » [1]
• Implications éthiques de l’analyse des mégadonnées – « Un certain nombre de questions sont soulevées concernant les nouvelles voies qui pourraient être nécessaires, les différentes approches réglementaires en Europe pour le traitement de ces données dans le cadre des directives européennes sur la protection des données à caractère personnel et les questions de qualité des données. S’ils ne sont pas correctement pris en compte par l’inclusion de la conception éthique dans la création de mégadonnées, ces problèmes éthiques pourraient devenir des facteurs limitatifs empêchant d’atteindre le plein potentiel. » [1]
[1] Vanderberg, O., Kozlakidis, Z., Schrenzel, J. et al (2018). « Le Contrôle des Maladies Infectieuses à l’ère de la Consolidation des Laboratoires Européens de Microbiologie Clinique: Nouveaux Défis et Opportunités pour le Patient et pour la Surveillance de la Santé Publique ». Frontières en médecine, 5 (15), pp. 1-7.
[2] Gebreyes, W. A., Dupouy-Camet, J., Newport, M. J., et al (2014). « The Global One Health Paradigm: Challenges and Opportunities for Tackling Infectious Diseases at the Human, Animal, and Environment Interface in Low-Resource Settings ». PLOS Negletected Tropical Diseases, 8 (11), e3257, pp. 1-7.
[3] Lippi, G et Plebani, M. (2020). « Le rôle critique de la médecine de laboratoire lors de la maladie à coronavirus 2019 (COVID-19) et d’autres épidémies virales « . Clin Chem Lab Med, 58(7), p. 1063 à 1069.
[4] Bhalla, N., Pan, Y., Farokh, A. (2020). « Opportunités et défis pour les Biocapteurs et des Outils d’Analyse à l’Échelle Nanométrique pour les Pandémies: COVID-19 « . ACS Nano, 14, p. 7783-7807.
[5] Armstrong, J., Rudkin, J. K., Allen, N et coll. (2020). « Dynamic linkage of COVID-19 test results between Public Health England’s Second Generation Surveillance System and UK Biobank ». Génomique microbienne, 6, pp. 1-9.
[6] Il s’agit de l’un des plus grands ouvrages de l’histoire de l’art. » Surveillance de la Santé Publique et Réglementation des utilisations significatives : Une crise d’opportunités « . Journal américain de santé publique, 102(3), pp. e1-e7
[7] Homolka, S., Pawlowski, L., Andres, S. (2020). « Deux Pandémies, Un Défi – Tirer Parti de la Capacité de Test Moléculaire des Laboratoires de tuberculose pour la Détection Rapide des Cas de COVID-19 ». Emerging Infectious Diseases, 26 (11), pp. 2549-2554.